Reconstrucción de Genomas a partir de Metagenomas (MAGs)
Last updated on 2025-04-06 | Edit this page
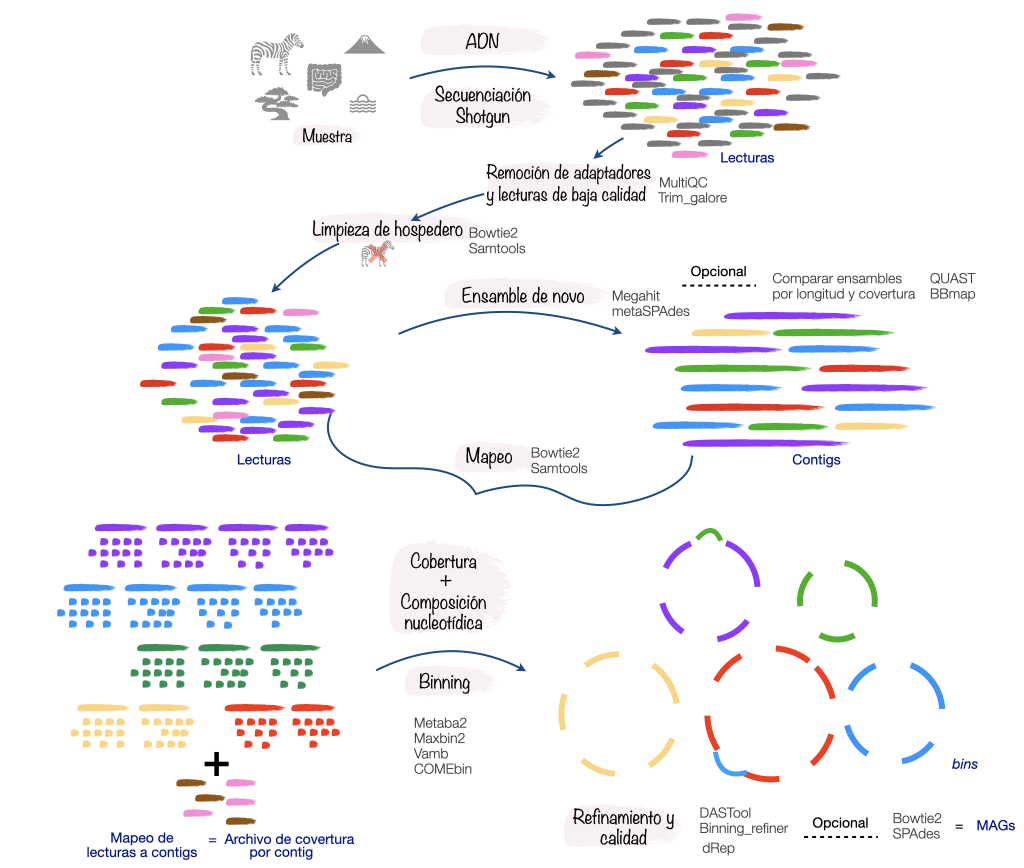
Genomas a partir de metagenomas
La metagenómica hace referencia al estudio de todo el ADN de los organismos que se encuentran en un ambiente. La secuenciación de este material genético produce lecturas que pueden ensamblarse para conocer la diversidad microbiana y sus funciones.
Típicamente los metagenomas pueden estudiarse mediante dos aproximaciones:
- La clasificación taxonómica de contigs o lecturas y la inferencia metabólica de los contigs.
- La reconstrucción de genomas a a partir de metagenomas (MAGs), clasificación taxonómica y la inferencia metabólica de los MAGs.
En este apartado nos enfocaremos en la segunda aproximación. Los
MAGs se reconstruyen a partir de un
ensamble metagenómico, los contigs de dicho ensamble se
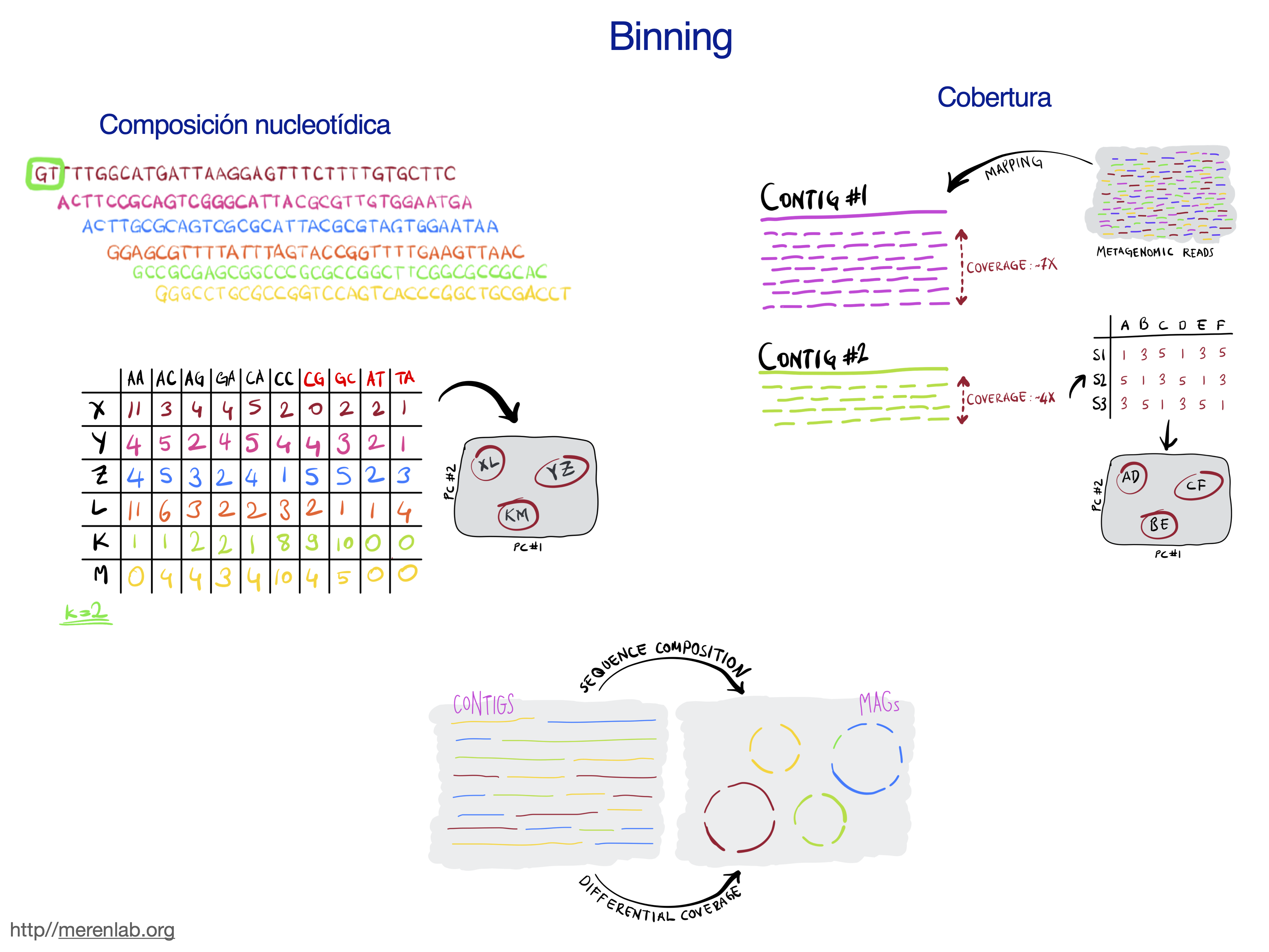
agrupan mediante la información de
cobertura y frecuencia de tetranucleótidos. Esta agrupación
puede generar errores, por lo que es indispensable evaluar la calidad de
los MAGs mediante la completitud y redundancia de genes de copia única
MerenLab y col.
Para obtener MAGs podemos seguir el siguiente flujo de análisis:
Ya que discutimos como seguir un flujo de análisis para reconstruir genomas entremos en acción, para ello analizaremos el metagenoma del pozol.
El pozol
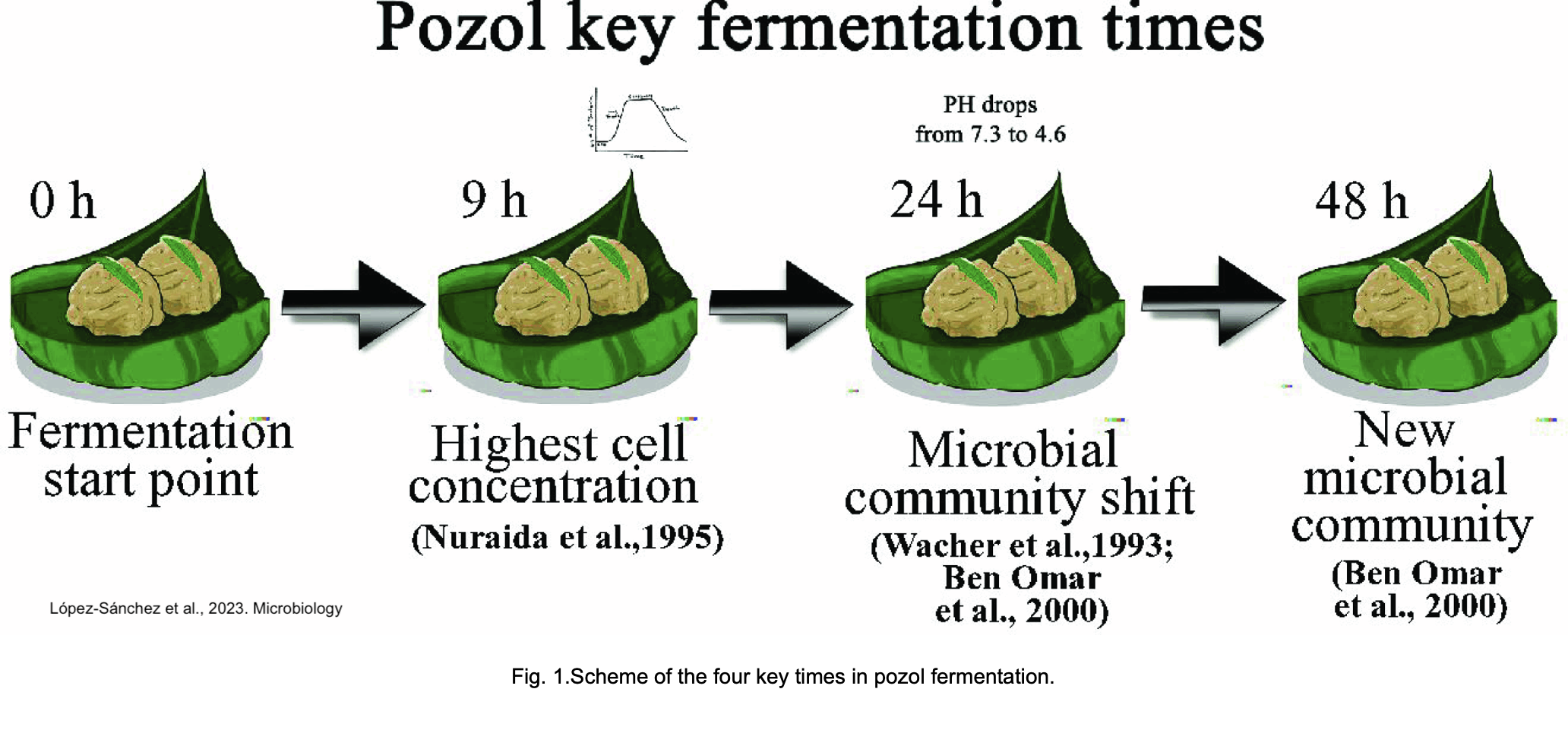
El pozol es un alimento ácido, fermentado a partir de maíz nixtamalizado, de importancia económica y cultural, se consume desde tiempos prehispánicos y se ha estudiado desde los años 50s.
Algunos puntos importantes que conocemos son:
* No se inocula y al final de su fermentación
tiene alta diversidad microbiana.
Es muy nutritivo, tiene un alto contenido de aminoácidos esenciales.
Es considerado como prebiótico, contiene fibras solubles y microorganismos benéficos para la salud intestinal humana.
Resolvamos preguntas biológicas mediante Metagenómica centrada en genomas
Imaginemos que se quiere impulsar la producción de esta bebida y para ello necesitan saber todo acerca de su naturaleza microbiana.
Una importante industria alimenticia los contacta como
expertos en ecología microbiana y les pide ayuda para
descubrir los siguientes puntos:
* ¿Qué actores microbianos están presentes
durante el proceso de fermentación?
* ¿Cómo ocurre la bioconversión
del maíz durante la fermentación, quién participa y cómo lo hace? ¿Qué
funciones metabólicas están ocurriendo?
* ¿Cambia la comunidad
microbiana a lo largo del proceso?
La empresa secuenció cuatro puntos de fermentación de muestras que se obtuvieron en un mercado de Tabasco. Las muestras se secuenciaron con Illumina NextSeq500 con lecturas pareadas de 75 pb. Los datos están públicos bajo el Bioproject: PRJNA648868
Limpieza de hospedero
Como las muestras contienen maíz, es indispensable remover las lecturas que correspondan a su genoma, no hacerlo producirá un ensamble muy fragmentado, mayoritariamente del maíz y poco microbiano. El autor del artículo amablemente nos proporcionó sus muestras libres del maíz y el código que usó para ello está disponible en un repositorio público de GitHub.
El artículo: López-Sánchez et al., 2023. Analysing the dynamics of the bacterial community in pozol, a Mexican fermented corn dough.
Espacio de trabajo
- Entra a tu cuenta en el servidor y sitúate en tu
$HOME - Obten los datos y la estructura de tu directorio del proyecto
- Entra al directorio del proyecto
BASH
# ve al $HOME
cd
# descarga
#wget https://zenodo.org/records/13911654/files/taller_metagenomica_pozol.tar.gz?download=1 -O taller_metagenomica_pozol.tar.gz
# descomprime
#tar -xvzf taller_metagenomica_pozol.tar.gz
# Entra al directorio del proyecto
cd taller_metagenomica_pozolSi en algún momento te pierdes entre directorios, puedes regresar al espacio principal asi:
Cómo vamos a trabajar durante el taller?
Reglas del juego
- En este tutorial haremos el ejemplo corriendo la muestra de 48 hrs.
- Se formaran 6 equipos (2 de los tiempos 0, 9 y 24 hrs).
- Los equipos discutirán y presentarán sus resultados cuando se indique en el tutorial.
La presente práctica sólo es una
representación del flujo de trabajo para el análisis metagenómico, sin
embargo, no sustituye los manuales de cada programa y el
flujo puede variar dependiendo del tipo de datos y pregunta de
investigación. De hecho para fines del taller, con frecuencia se
utilizan las lineas de comando más simples para eficientar tiempo y
recursos, tómalo en cuenta.
Cada programa tiene una ayuda y un manual de usuario, es
importante revisarlo y conocer cada parámetro que se
ejecute. En terminal se puede consultar el manual con el comando
man y también se puede consultar la ayuda con
-h o --help, por ejemplo
fastqc -h.
Para tenerlo presente
En bioinformática cualquier línea de comandos generará un resultado, de ahí a que esos resultados sean correctos puede haber una gran diferencia. En cada paso detente a revisar la información de cada programa, lee el manual, visita foros de ayuda y selecciona los argumentos que se ajusten a las necesidades de tus datos.